Je me permets de poster ici car j'ai un problème qui (je pense) relève de la prise en main du logiciel et de la manipulation des données, plus que d'une compréhension statistiques. Je précise que je suis débutante, et que l'utilisation du logiciel R est imposé par ma formation (je ne peux pas passer par un autre logiciel ou autre méthode).
J'ai un tableau de donnés avec 10 colonnes, dont les premières sont abondance et richesse spécifique d'espèces (remplies de 7 à 250), puis les 8 autres sont des types de milieux, remplies avec des % (de 0 à 1).
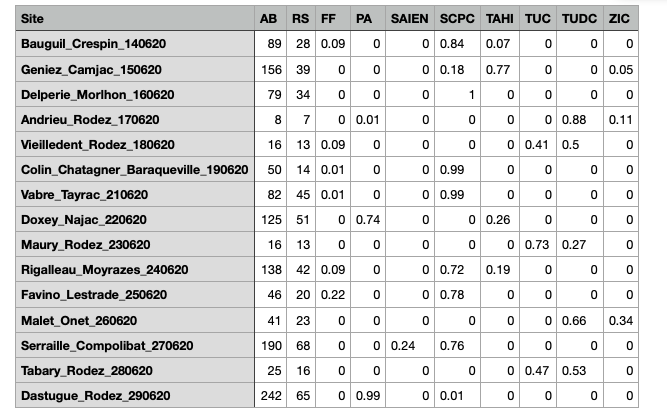
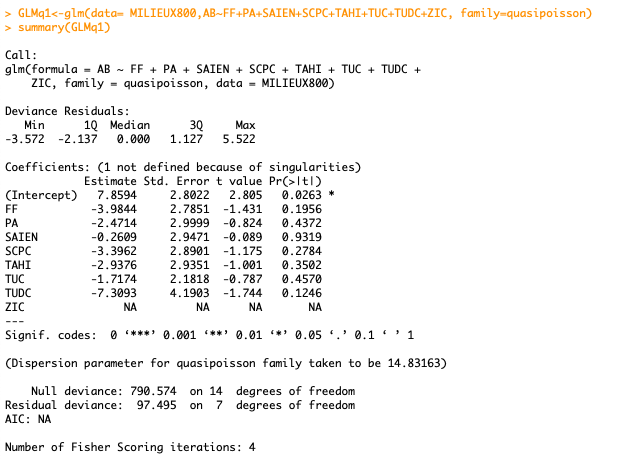
J'ai effectué une GLM de mon abondance et de ma richesse avec ces types (quasipoisson car il y avait surdispersion) :
L'un de mes types de milieux (le dernier) ressort en "NA" dans ma GLM, alors que toutes les cases sont remplies et qu'il n'y a aucun caractère spécial. Devant "Coefficients", il y a un message "1 not defined because of singularities", ce qui pourrait vouloir dire que ma variable en question est trop corrélée aux autres, mais quand je fais cor(), ce n'est pas le cas.
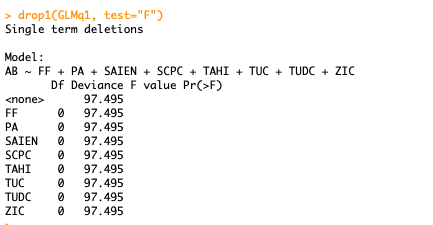
Lorsque j'ignore le NA et que je continue ma GLM jusqu'à ma fonction drop1, le tableau donne la même déviance pour tous mes milieux, et sans pvalue.
Lorsque je retire cette variable "problématique", tout marche.
1) Y'a t il une explication au fait que ma variable apparaissent en NA et ne veuille pas se lire ?
2) Si non, est-ce que supprimer cette variable va altérer mon modèle ?
Je vous remercie par avance pour votre aide,
Louise