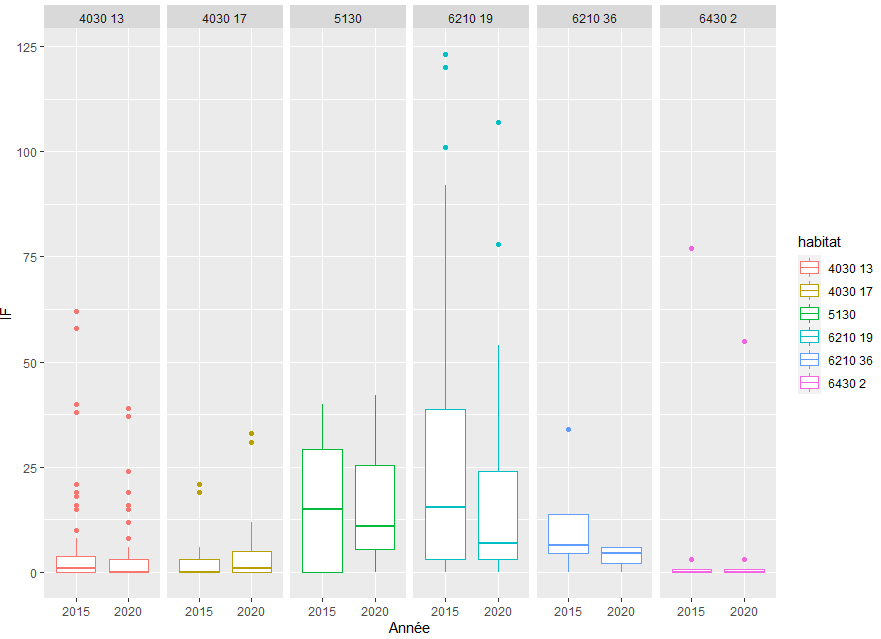
Je suis débutante sur R, j'ai réalisé un ggplot avec plusieurs données, afin de voir à la fois une évolution entre les années, et entre mes habitats, voici ma ligne de code et le graphique obtenu :
ggplot(habitatIF)+geom_boxplot(aes(x="2015",y=IF_note_2015,color=habitat))+geom_boxplot(aes(x="2020",y=IF_note_2020,color=habitat))+facet_grid(~habitat)+labs(x="Année",y="IF")
habitat IF_note_2015 IF_note_2020
6210 19 39 54
6210 19 24 37
4030 13 0
J'ai réalisé un pairwise.wilcox.test en amont afin de déterminer les différences significatives. Je souhaite maintenant annoter chaque boxplot avec des lettres pour montrer la significativité entre les différents éléments, cependant je n'arrive pas à annoter un seul boxplot à la fois, avec la fonction annotate, lorsque je met annoter x1, il annote tout les boxplot en 2015. Voici ce que je souhaite obtenir (ajouter sur paint) :
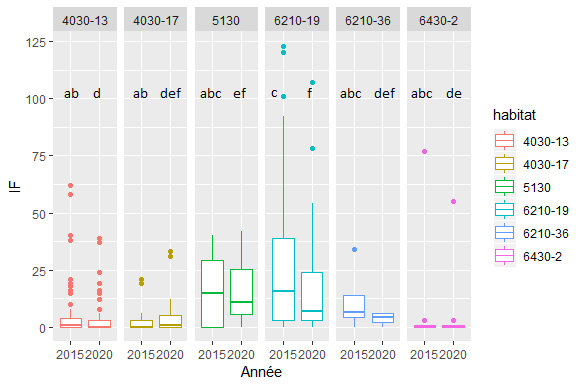
Comment puis je procéder ?
Merci de vos réponses